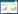文章信息
- 董倩, 李彩霞, 赵蕾, 郭瑜鑫, 马温华, 孙辉, 叶健, 李万水, 朱波峰, 孙敬
- DONG Qian, LI Caixia, ZHAO Lei, GUO Yuxin, MA Wenhua, SUN Hui, YE Jian, LI Wanshui, ZHU Bofeng, SUN Jing
- 法医DNA快速检验研究进展
- Advances in forensic DNA rapid test
- 中国测试, 2017, 43(7): 59-65, 71
- China Measurement & Test, 2017, 43(7): 59-65, 71
- http://dx.doi.org/10.11857/j.issn.1674-5124.2017.07.012
-
文章历史
- 收稿日期: 2016-08-10
- 收到修改稿日期: 2016-10-20
2. 公安部物证鉴定中心 北京市现场物证检验工程技术研究中心 现场物证溯源技术国家工程实验室, 北京 100038
2. A Key Laboratory of Forensic Genetics, Beijing Engineering Rersearch Center of Crime Scene Evidence Examination, National Engineering Laboratory for Forensic Science, Institute of Forensic Science, Beijing 100038, China
DNA分型在法医学,亲子鉴定,基因型分析等方面应用广泛[1-2]。常规的法医DNA分型通常包括DNA的提取、定量、扩增、纯化、片段的分离和检测,需要8~10 h,已无法满足现有的需求。近年来国内外均通过各种方法来尽可能缩短DNA分型时间。本文就国内外法医DNA-STR快速检验的研究进展和成果,分别从快速提取、快速扩增、快速设备、快速试剂与快速设备相结合4个方面对有关DNA快速检验的技术方法,实验原理进行说明和举例论证,并对该行业在快速检验方法的选择上提出明确的建议。
1 生物检材快速提取的研究现状DNA分型中,一般接触性的检材样本DNA的提取通常要1 h以上[3],而且会因提取样本差异,面临诸多挑战以及花费更长时间。在样本提取的载体方面,法医DNA最常用的载体是棉签或者拭纸,它们共同点是对DNA的吸附性强,但损害大,且分布不均一。早在2007年孙海汐等[4]发现了一种微量DNA的快速检验技术:生理盐水簌口法,可快速获取足以满足PCR扩增的DNA,并且便宜、方便;而棉签取材不稳定,有一定吸附性,损害大,DNA含量少,牙签效果则更差。在样本提取所需环境方面,使用碱性裂解法可在5~10 min内快速提取常规的生物检材,2010年袁自闯等[5]采用差异裂解法,同时结合脱氧核苷酸核酶DNase Ⅰ和碱性裂解法,调整溶液各成分的浓度及pH值,使用改良的碱性裂解液可在8 min内完成各类生物物证的提取,实现了快速获得含有精斑的混合斑中的精子DNA。在样本提取的试剂方面,Chelex 100最为常见,可用于全血、混合斑、毛发检材,并在数分钟内即可完成提取,但DNA纯度不高。对于DNA含量少的检材,通常采取M48磁珠法或是硅珠方法提取,2013年高林林等[6]利用PrepFiler Express BTA提取试剂盒结合AutoMate自动化法医DNA提取工作站在3 h内实现了对骨粉和牙粉的裂解以及纯化。在法医DNA样本提取的设备方面,一般体液样本通常利用离心机分离DNA分子,或通过微型过滤器从流体样本中抽提,需要20~30 min才能完成,并在提取过程中使用过多的有毒化合物。现今最新设备为2013年华盛顿大学的工程师联合NanoFacture公司研发出的新型DNA提取设备——侵入流体样本(唾液、痰液或血液)的显微探针,通过在液体中施加电场,微型探针在其表面吸附DNA大小的颗粒,较大颗粒因击中探针尖端而被弹开。采用这一技术,在3 min内就可分离和提纯DNA分子。Ma C[7]用该显微探针分别对4个不同来源样本,包括细菌、酵母、人血和病毒进行了提取和比较,均可以获得最大产量DNA,并进一步验证了该新型显微探针避开了传统方法所有步骤,以更高效、环境更友好的简单方式从流体样本中提取DNA。
2 快速PCR扩增检验快速PCR是指在保证PCR扩增反应结果准确性的前提下,尽可能地缩短PCR反应时间。国内外已经研发出各种商品化的试剂盒不同程度地缩短了PCR扩增时间。目前快速PCR的研究主要从对DNA聚合酶的改进、对缓冲体系(Buffer,Mix)的优化和改进以及结合快速扩增程序等方面来实现,主要表现为免提取的扩增检验和快速扩增检验两方面。
2.1 免提取的扩增检验免提取扩增又称直接PCR。它是指将未经过处理的生物样本直接加入PCR反应体系并获得扩增产物[8]。免提取扩增检验针对的样本是血液、血卡、口腔拭纸、唾液卡、棉签、毛发根部等常规检材,对于混合检材和疑难降解检材目前没有研究。国内外早已有对全血DNA免提取而直接扩增的研究,Mercier等[9]在1990年就报道了全血扩增和以全血样本提取DNA扩增的检测具有同等效果。已上市的商品化直扩试剂盒针对的样本均是血卡或是唾液卡。这些直扩试剂盒虽然会有位点选取和数目的差异,个体识别能力也不尽相同,并且对人群具有一定的选择适用性,但相同点均是通过各自专用的酶及其配套的缓冲体系联合相应扩增程序来实现快速检测(见表 1[10])。2013年Flores S报道的GloberFiler Express[11]采用更强效的抗抑制缓冲体系和专用酶,同时结合优化的热循环参数将PCR扩增时间缩短至40 min。Myers等[12]将PowerPlex®8D和Identifiler® Direct进行比较,虽然两种直扩试剂盒的性能不一样,但通过改变样本DNA的量,调整循环参数和适当延长或减少毛细管进样时间,最终使两种试剂盒对同一样本的检测结果一致且分型准确。
2.2 快速扩增检验
一种常用的快速扩增研究方法是选用合适的快速酶并结合快速循环程序对现有的常规试剂盒进一步优化,以达到缩短PCR扩增时间为目的。如2008年Vallone等[13]将PyroStart和SpeedSTAR两种酶结合使用,PyroStart酶(Fermentas,Glen Burnie,MD)可实现快速扩增,SpeedSTAR酶(Takara Bio USA,Madison,WI)可提高腺苷酸化,优化各位点间的峰平衡,同时结合快速循环程序将Identifiler®扩增时间缩短为36 min;Amanda Foster[14]选择SpeedSTARTMHS DNA聚合酶,使用Bio-Rad C1000TM扩增仪进一步提高升降温循环的速率,同时修正每步的热循环参数,并采用两步代替3步的PCR方法使AmpFLSTR® Identifiler®整个扩增的时间为26 min;Bahlmann等[15]通过将PowerPlex®S5试剂与分别与3种可替代酶相结合研究,并同时使用快速循环程序将PCR的扩增缩短为1 h;James White[16]通过使用快速启动酶Speed STARTMHS Polymerase,结合Veriti®(Applied Biosystems,Foster City,CA,USA)rapid thermal cycler的快速循环程序,将PowerPlex® 16 HS扩增时间缩短为1 h。另一种研究方法是将常规试剂盒与快速试剂盒相结合,或是将两种或多种快速试剂联合应用。如王鸿迪等[17]联合运用AmpFLSTR Sinofile试剂盒,QIAGENFast Cycling PCR试剂盒将扩增时间缩短至60 min以内。刘琳等[18]在2013年将AmpFLSTR®Identifiler®分别与4种不同的快速PCR检测试剂——TaKaRa,Fermentas,Invitrogen,Thermo联合应用初次构建快速PCR扩增检验,并于2014年将AmpFLSTR®Identifiler Plus试剂盒与TaKaRa快速PCR检测试剂联合使用,从而将常规的扩增时间175 min缩短为为55 min,并再次验证了将TyperTM19试剂盒与快速试剂结合可有效缩短PCR扩增时间。
由此可见,多种方法和途径可以将常规PCR的扩增时间(3~5 h)不同程度的缩短,所以可以使用国产试剂结合多种商品化的试剂盒中的酶或国产的酶结合多种商品化的试剂,或是采用多种高保真、强抗抑制力的酶与多种抑制能力更强的缓冲液(均从已上市的多种试剂盒中可以获得)相互结合,多次比较,通过微调扩增体系各成分的比例、浓度,控制添加剂、抑制剂等措施来实现不同程度的快速扩增。
3 快速设备实现法医DNA快速检验设备主要包括快速扩增设备,快速电泳分析设备。
3.1 快速扩增设备不同扩增设备的性能,效率明显不同。Phillip Belgrader[19]最初在1998年报道使用了比9700扩增仪具有更快升降温速率和更好温控性能的微型装置充电型热循环仪器MATCI,可在21 min内完成PCR扩增。Amanda Foster[14]在建立快速PCR的方法时,将SpeedSTARTMHS DNA聚合酶分别与Bio-Rad C1000TM,Eppendorf Mastercycler®,Finnzymes Piko®扩增仪器联合使用,最终选用Bio-RadC1000TM扩增仪提高升降温循环的速率,使AmpFLSTR®Identifiler®整个时间为26 min。所以尽可能选用升降温速率快,温控性能好的快速PCR,如Bio-Rad C1000TM,Eriti扩增仪(美国AB公司),LifePro扩增仪(杭州博日科技有限公司)等可以进一步有效缩短检测时间。
3.2 快速电泳分析设备目前国内实验室普遍配置的快速电泳检测设备有3130XL型遗传分析仪,可在45 min完成16个样本的同时检测,3730型或3730XL型遗传分析仪一次可同时完成48或96个样本检测,新型的3500XL型遗传分析仪可在30 min内完成24个样本的同时检测,但是这些检测设备仍然滞后于当前大批量生物物证检验和当代法庭科学的需求。对快速电泳分析方面的研究,2011年平原等[20]用6+1 STR试剂盒结合一种新型毛细管电泳凝胶EX-Q20进行快速电泳分析,与SinofilerTM试剂盒结合POP4胶进行比较,6+1 STR试剂盒结合新型毛细管电泳凝胶能够得到全部分型结果且耗时较短。在快速电泳部,该新型毛细管电泳凝胶EX-Q20合成丙烯酰胺-N,N-二甲丙基烯酰胺,形成一种新型准互穿聚合物网络,解决了传统毛细管电泳凝胶不能兼顾分离效果与自涂敷功能的难题,同时通过局部改良和电泳参数的设置可节约成本。
4 微流控芯片技术 4.1 微流控芯片技术在法医DNA领域的发展现状微流控芯片技术又称微全分析系统(miniaturized total analytical system,μTAS),是把生物、化学、医学分析过程的样品制备、反应、分离、检测等基本操作单元集成到一块微米尺度的芯片上,自动完成分析全过程。由于它在生物、化学、医学等领域的巨大潜力,已经发展成为一个生物、化学、医学、流体、电子、材料、机械等学科交叉的崭新研究领域。近年来诸多学者基于微流控芯片技术的角度,对涉及快速检验芯片DNA提取、芯片PCR扩增、毛细管芯片电泳及集成式芯片均进行了深入的研究。
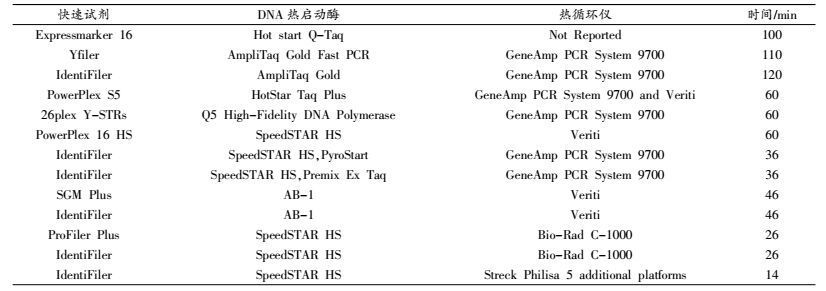
在芯片DNA提取技术方面,国内研究最多的是以二氧化硅微球为载体的固相提取,它是M48磁珠法和硅珠法提取DNA的微型化。原理如下:以二氧化硅微球、磁性微球等介质作为DNA的固相载体,首先采用高离子盐液进行DNA吸附、有机溶剂洗脱,最后使用低离子缓冲液洗脱DNA。如2010年Duarte[21]报道了基于固相萃取原理的玻璃材质DNA提取芯片,如图 1所示,腔室的大小1.5 cm×1.4 mm×200 μm,以确保预提取的DNA溶液量恒定;在磁场作用下整个腔室充满磁性微球和盐酸胍(GuHCl),整个装置完成DNA的提取和纯化。Cady等[22]也报道了基于固相萃取原理的DNA提取芯片,硅珠、sol-gel等固相载体填充于芯片中,实现了血样等的DNA提纯。陈兴等[23]验证了在多孔氧化硅芯片和普通芯片上进行DNA提取实验,多孔氧化硅芯片提取DNA的效率更高。目前各种聚合物基底如聚甲基丙烯酸甲酯(PMMA)、聚碳酸酯(PC)等由于价格低廉、加工方便已经被成功用于DNA提取系统。如2007年刘惯超等[24]制作的塑料基(PMMA)的DNA提取芯片表明:经过溶胶涂渍、在内表面制作二氧化硅层的芯片能够实现从血液中提取DNA。王聿佶等[25]采用PDMS(聚二甲基硅氧烷)芯片,能对生物样品中的DNA进行有效快速提取,并且在30 min内完成PCR产物的提纯。国外还有报道基于pH诱导的静电吸附DNA提取,基于UV活化的聚碳酸酯表面的DNA提取[26],基于纳米多孔过滤膜的DNA提取[27]。
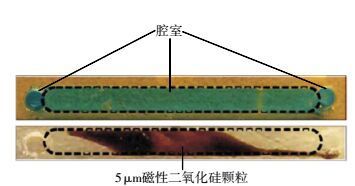
芯片PCR扩增与普通PCR扩增仪相比,扩增速度有明显优势。普通台式PCR扩增仪的产物扩增管升降温度总是远远滞后于扩增仪腔本身的温度,而芯片PCR则具有明显优势。Heidi Giese[28]报导对比普通台式PCR仪器和微流控芯片使用SpeedSTAR试剂对样本进行扩增,图 2为显示的结果,普通台式PCR仪器扩增所用时间为145.1 min,芯片PCR的扩增所用时间为19.6 min,远远快于普通台式PCR仪器。采用升降温速率性能佳的PCR扩增仪可以明显缩短PCR的扩增时间,同样,提高芯片温度控制是实现芯片PCR快速扩增的关键,代表性的研究有Hagan等[29]以非接触的红外加热方式进行热循环扩增,结合SpeedSTARTDNA聚合酶,仅在45 min内完成就完成了16个STR位点在微芯片上的扩增。Mathies小组[30]则以接触式温度控制方式实现芯片上PCR的快速扩增,同时证明了芯片PCR-CE的可行性。此外微机电技术和温度传感器等方面的研究也证明了可以缩短芯片PCR扩增时间。
芯片毛细管电泳具有进样量少,灵敏度高,分析速度快等特点,非常适合法医DNA-STR的快速检验。毛细管电泳芯片由于尺寸小,可施加较大场强,所以在几秒钟内就可完成对样品的分离,微阵列毛细管电泳芯片可实现高通量检测则成为目前学者研究的热点。2002年Emrich CA等[31]报道的将高通量384孔毛细管阵列微电泳芯片集成在半径仅为8 cm的圆盘上,7 min内实现了同时对384份样品的检测;2006年Yeung等[32]制作的高通量96孔毛细管阵列微电泳芯片可在30 min内同时实现96个样本检验,这些均进一步显示了微芯片技术在DNA-STR快速分析方面的巨大潜力和优势。为了防止样品间发生交叉污染,降低成本,可以构建一次性的塑料材质的微整列电泳平台。
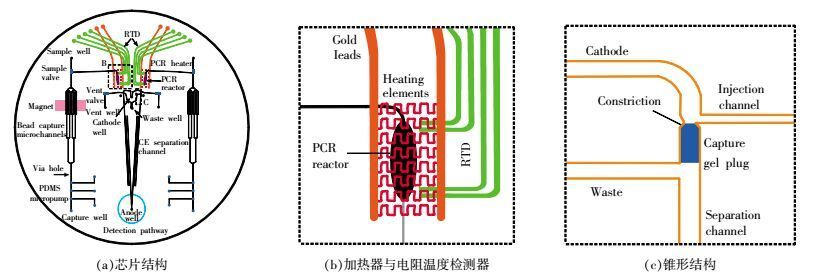
集成式芯片是当前微流控芯片研究的核心,如Hagan等[29]将DNA提取和RCR扩增集于一块芯片上,通过采用固相提取,使用快速启动DNA聚合酶及红外加热方式,在45 min内完成了口腔拭子的提取和PCR扩增;Roux等[33]将芯片PCR扩增和电泳检测集成在一次性的塑料芯片上,通过非接触式红外加热方法仅在7 cm芯片上完成了电泳分离;季旭等[34]研制的一种集成PCR反应室与毛细管电泳CE的生物芯片,将硅片上制造的通过电阻加热的凹槽作为扩增池,在扩增池下游设计制造毛细管电泳系统,并通过紫外光等检测器判断结果,从而实现了样品扩增、电泳分离和紫外光检测的单片集成。Liu等[35]将特定DNA模板的纯化、PCR扩增、进样以及电泳全集成于一张微流控芯片上,并可在3 h内实现对样本DNA的检验(如图 3所示),该全集成芯片原理结构为两个完全相同的分析系统在4 in(1 in=2.54 cm)玻璃晶片上形成对称的双峰,每个分析系统包括聚二甲基硅氧烷(PDMS)微泵和两个PDMS微型阀用于流体控制,首先通过4 cm长的珠捕获结构用于DNA分叉通道系统模板的捕获。图 3(b)为加热器和电阻温度检测器(RTD),用于控制250 nL PCR室的扩增。图 3(c)为500 mm长的双T通道锥形结构,用于控制PCR的纯化和进样,24 cm和14 cm长的通道用于CE分离,同时这两个系统共享阳极、阴极,进一步节省了芯片的使用空间。文献[36]在2014年将全集成芯片技术成功应用于对模拟犯罪现场样本检材的检验。全过程包括制备特殊酶的反应液用于DNA的提取,采用红外非接触式控温技术完成PCR扩增和高分辨的电泳分离方法在2 h内完成了对样本的准确分型,并与传统方法分型结构一致。
4.2 芯片化法医DNA检测的实现方式 4.2.1 微流控芯片DNA提取条件的优化
对提取进液量进行筛选,包括提取过程中的水,NaOH,HCL以及最后冲洗的水和TE缓冲液的体积,通过控制变量法,分别设置不同的体积梯度和停留时间,进行自动化提取,按照正常的扩增体系,并同时设置阳性对照和空白对照进行比较确定出最佳进液量;对于提取进液的管道可以设置多种不同的长度并运行自动化提取流程,提取后取出芯片上的载体物质进行常规的PCR扩增并检测,通过比较不同长度管道的扩增效果确定出最佳管道长度。
4.2.2 微流控芯片PCR扩增与优化对芯片PCR材料进行生物相容性验证是芯片PCR正常扩增的前提,因为一般芯片的结构是两种以上的材料通过键和组成,在此过程中可能会有抑制物释放出来从而影响后续的PCR扩增。直接将芯片的原材料剪取小块,放入扩增体系,加阳性标准品,扩增检测,同时设立不添加原材料的阳性对照,3个重复;分别用高温浸泡的芯片材料和去离子水配置PCR反应体系进行扩增检测,同时设置正常去离子水为阳性对照,3个重复;用去离子水冲洗键合芯片材料3次,配置PCR反应体系进行扩增检测,同时设置正常去离子水为阳性对照,3个重复;对提取装置最后的冲洗水进行研究,配置PCR反应体系进行扩增检测,同时设置正常去离子水为阳性对照,3个重复,对以上结果进行比较,如果荧光信号基本无差异则相容性良好。
微流控芯片PCR的实现重点在于对芯片材料属性的研究和芯片扩增体系的优化两部分。首先了解芯片的基本结构,一般芯片腔室的实际温度和PCR扩增仪显示的温度有较大差别,所以需进行芯片温度校准,以保证规定时间内有足够的退火温度和延伸时间;由于芯片材料的不同,即使对芯片PCR没有抑制作用,但是由于芯片比表面系数的增加,对蛋白酶的吸附逐渐增加,以及载体的存在,吸附会更明显,所以通常对芯片进行预处理,使内壁和反应体系隔绝,可以采用一定浓度的BSA、PEG进行芯片涂布预处理。微流控芯片对PCR某些成分的吸附,会影响PCR的扩增效果,通过对扩增体系中不同反应液的分析和浓度调节,尽量改善扩增效果。采用控制变量法,分别对不同成分设置浓度梯度,进行扩增检测,并且同时设立常规检测进行比较;操作者可在扩增体系中加入不同浓度梯度的BSA、PEG,并设立常规阳性对照,3个重复进行比较,从而确定出最佳扩增体系。
4.2.3 毛细管芯片电泳系统的优化根据毛细管电泳技术的基本原理,影响电泳的因素主要有电场强度、毛细管的材料和内壁、缓冲液的成分、溶液的pH值和浓度、温度等。对于电泳筛分介质的制备,参考已有成果制作筛分介质并同时选择3种商品化的筛分介质进行比较;对于管道表面修饰,参考已有研究成果进行动态涂布、静态涂布处理,填充自制的筛分介质后备用。芯片毛细管电泳实验采用悬浮式的进样方式,参考已报道的研究成果,选取合适的电压、时间、光信号进行电泳检测。
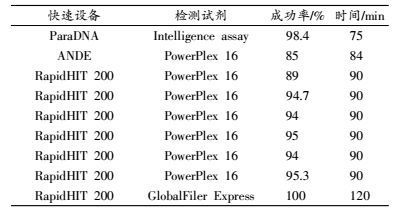
5 全自动DNA分型检测设备近几年,国外相继有3种全自动DNA分型检测设备上市(见表 2)。2013年美国IntegenX公司全球首家推出RapidHIT200 DNA快速检测仪,针对唾液、血液、血斑、唾液斑等样品,通过选用Promega PowerPlex 16HS试剂,分型成功率均在89%以上,并且可在现场90 min内快速完成DNA检测[37-38],并且输出的结果可与CODIS及中国DNA数据库比对。2016年最新升级的RapidHIT200 DNA快速检测仪与GlobalFiler Express试剂相结合,可同时检测7个样本。文献[39]用150份口腔样本进行验证,进一步说明了检测准确率更高,设备灵敏度高(从6 260个细胞中获得37.5 ng DNA)。DNAScanTM快速检测设备是美国多政府部门合作在法律部门,刑事实验和生物测定实验室推行快速DNA检测技术项目(Acceleted Nuclear Equipment,ANDE)研究成果之一。该快速设备使用一体化BioChipset试剂盒,在85 min内同时完成5个单一来源样本的DNA检测,符合美军用810F耐用标准,并且通过冲击试验、振动试验、坠落实验及环境变化检测实验均证实该设备经久耐用。Tan E等[40]对该仪器设备的性能,检测的灵敏度等进行了进一步的验证。2014年,LGC法医刑侦科学协会成功研发的ParaDNA Screening快速检验系统可在75 min内同时实现对4个样本的DNA检测。
6 结束语
综上,缩短对样本DNA提取,扩增,纯化,电泳检测中任意环节所消耗的时间均可实现不同程度的法医DNA的快速检验。国内外成熟的方法是使用商品化的提取试剂,快速试剂,或是通过将快速试剂与性能好的快速扩增设备,电泳设备相结合来实现。但是本文认为直接PCR会因为样本中各种潜在的未知抑制物影响酶的活性,缺乏定量步骤会影响DNA的分型结果,所以使用商业化直扩试剂盒时,要严格按照试剂盒所规定的样本量进行实验。目前DNA-STR快速检验主要适用于常规检材,但对于一些疑难检材,混合样品,以及高度降解的检材的使用仍然是个严峻的挑战,但是要明确:快速检验的目的主要是协助公检法处理一些棘手和特殊的案件,如处理案件性质恶劣但必需在极其有限时间需要得到分型结果。虽然国外商业化的3种便携式的全自动快速检验仪器已经上市,但普遍存在的问题是检测成本过高,并且不能自主选择样本数,国内普及困难。全集成微流控芯片技术是目前快速检验研究的核心,即使芯片的适用具有一定的样本选择性,并且芯片结构复杂,成本高,但是国内的研究面临的主要瓶颈是如何开发配套的软件来分析自制的微芯片的电泳结果。法医DNA快速检验需从检测样本数目的自主选择、再检测样本能力以及疑难,混合样本的角度出发,研制出我国国产的便携式全自动快速检测仪器,并实现推广普及。
| [1] | CHAKRABORTY R, STIVERS D N, SU B, et al. The utility of short tandem repeat loci beyond human identification:implications for development of new DNA typing systems[J]. Electrophoresis, 1999, 20(8): 1682. DOI:10.1002/(ISSN)1522-2683 |
| [2] | DANI S U, GOMES-RUIZ A C, DANI M A. Evaluation of a method for high yield purification of largely intact mitochondrial DNA from human placentae[J]. Genetics & Molecular Research Gmr, 2003, 2(2): 178–84. |
| [3] | 骆继怀, 陈晓军, 孙红兵, 等. 接触性生物检材提取DNA方法的比较[J]. 兰州大学学报(医学版), 2015, 41(1): 18–22. |
| [4] | 孙海汐, 蔡金挺, 朱琳楠, 等. 一种改良的人DNA微量快速检验技术[J]. 中国实验诊断学, 2007, 11(6): 788–790. |
| [5] | 袁自闯, 金洪年, 赖跃, 等. DNase-I纯化结合碱性裂解法提取混合斑精子DNA[J]. 中国法医学杂志, 2010, 25(1): 10–12. |
| [6] | 高林林, 徐念来, 谢炜, 等. 利用AutoMateExpressTM自动化法医DNA提取系统提取骨骼及牙齿DNA[J]. 法医学杂志, 2013, 29(2): 127–129. |
| [7] | MA C, LI C, WANG F, et al. Magnetic nanoparticles-based extraction and verification of nucleic acids from different sources[J]. Journal of Biomedical Nanotechnology, 2013(9): 703–709. |
| [8] | LINACRE A, PEKAREK V, SWARAN Y C, et al. Generation of DNA profiles from fabrics without DNA extraction[J]. Forensic Science International Genetics, 2010, 4(2): 137–141. DOI:10.1016/j.fsigen.2009.07.006 |
| [9] | MERCIER B, GAUCHER C, FEUGEAS O, et al. Direct PCR from whole blood, without DNA extraction[J]. Nucleic Acids Research, 1990(18): 5908. |
| [10] | ROMSOS E L, VALLONE P M. Rapid PCR of STR markers:applications to human identification[J]. Forensic Science International Genetics, 2015(18): 90–99. |
| [11] | FLORES S, SUN J, KING J, et al. Internal validation of the globalFiler express PCR amplification kit for the direct amplification of reference DNA samples on a high-throughput automated workflow[J]. Forensic Science International Genetics, 2014(10): 33–39. |
| [12] | MYERS B A, KING J L, BUDOWLE B. Evaluation and comparative analysis of direct amplification of STRs using PowerPlexR 18D and IdentifilerR direct systems[J]. Forensic Science International Genetics, 2012, 6(5): 640–645. DOI:10.1016/j.fsigen.2012.02.005 |
| [13] | VALLONE P M, HILL C R, BUTLER J M. Demonstration of rapid multiplex PCR amplification involving 16 genetic loci[J]. Forensic Science International Genetics, 2008, 3(1): 42–45. DOI:10.1016/j.fsigen.2008.09.005 |
| [14] | FOSTER A, LAURIN N. Development of a fast PCR protocol enabling rapid generation of AmpFLSTRR, IdentifilerR, profiles for genotyping of human DNA[J]. Investigative Genetics, 2012, 3(1): 1–11. DOI:10.1186/2041-2223-3-1 |
| [15] | BAHLMANN S, HUGHES-STAMM S, GANGITANO D. Development and evaluation of a rapid PCR method for the PowerPlexR; S5 system for forensic DNA profiling[J]. Legal Medicine, 2014, 16(4): 227–233. DOI:10.1016/j.legalmed.2014.04.003 |
| [16] | WHITE J, HUGHESSTAMM S, GANGITANO D. De-velopment and validation of a rapid PCR method for the PowerPlexR 16 HS system for forensic DNA identification[J]. International Journal of Legal Medicine, 2014, 129(4): 1–9. |
| [17] | 王鸿迪, 董海成, 于俊峰, 等. AmpFISTR Sinofiler快速PCR扩增初探[J]. 中国法医学杂志, 2012, 27(5): 397–398. |
| [18] | 刘琳, 项林平, 郭磊, 等. 4种快速PCR检测试剂法医学应用的初步研究[J]. 生命科学研究, 2013, 17(5): 431–435. |
| [19] | BELGRADER P, SMITH J K, WEEDN V W, et al. Rapid PCR for identity testing using a battery-powered miniature thermal cycler[J]. Journal of Forensic Sciences, 1998, 43(2): 315–319. |
| [20] | 平原, 周怀谷, 许炎, 等. 6+1 STR试剂盒和电泳凝胶EX-Q20用于DNA快速检验[J]. 法医学杂志, 2011, 27(6): 444–446. |
| [21] | DUARTE G R, PRICE C W, LITTLEWOOD J L, et al. Characterization of dynamic solid phase DNA extraction from blood with magnetically controlled silica beads[J]. Analyst, 2010, 135(3): 531–537. DOI:10.1039/b918996c |
| [22] | CADY N C, STELICK S, BATT C A. Nucleic acid pur-ification using microfabricated silicon structures[J]. Biosensors & Bioelectronics, 2003, 19(1): 59–66. |
| [23] | 陈兴, 崔大付, 刘长春, 等. 多孔氧化硅微流控DNA提取芯片的研制[C]//全国微全分析系统学术会议, 2005. |
| [24] | 刘惯超, 肖宏, 李荔, 等. 溶胶-凝胶法制作塑料基DNA提取芯片以及应用于从人血中提取DNA[J]. 材料科学与工程学报, 2007, 25(1): 122–124. |
| [25] | 王聿佶, 陈翔, 曹慧敏, 等. 一种新型微流体DNA提取芯片的研究[J]. 微纳电子技术, 2007, 44(9): 853–856. |
| [26] | MALGORZATA A W, LLOPIS S D, WHEATLEY A, et al. Purification and preconcentration of genomic DNA from whole cell lysates using photoactivated polycarbonate(PPC) microfluidic chips[J]. 2006, 34(10):74. |
| [27] | KIM J, GALE B K. Quantitative and qualitative analysis of a microfluidic DNA extraction system using a nanoporous AlO(x) membrane[J]. Lab on A Chip, 2008, 8(9): 1516–1523. DOI:10.1039/b804624g |
| [28] | GIESE H, LAM R, SELDEN R, et al. Fast multiplexed polymerase chain reaction for conventional and microfluidic short tandem repeat analysis[J]. Journal of Forensic Sciences, 2009, 54(6): 1287–1296. DOI:10.1111/jfo.2009.54.issue-6 |
| [29] | HAGAN K A, REEDY C R, BIENVENUE J M, et al. A valveless microfluidic device for integrated solid phase extraction and polymerase chain reaction for short tandem repeat(STR) analysis[J]. Analyst, 2011, 136(9): 1928–1937. DOI:10.1039/c0an00922a |
| [30] | LAGALLY E T, MEDINTZ I, MATHIES R A. Single-molecule DNA amplification and analysis in an integrated microfluidic device[J]. Analytical Chemistry, 2001, 73(3): 565–570. DOI:10.1021/ac001026b |
| [31] | EMRICH C A, TIAN H, MEDINTZ I L, et al. Micro-fabricated 384-lane capillary array electrophoresis bioanalyzer for ultrahigh-throughput genetic analysis[J]. 2002, 74(19):5076-5083. |
| [32] | YEUNG S H, GREENSPOON S A, MCGUCKIAN A, et al. Rapid and high-throughput forensic short tandem repeat typing using a 96-lane microfabricated capillary array electrophoresis microdevice[J]. Journal of Forensic Sciences, 2006, 51(4): 740–747. DOI:10.1111/jfo.2006.51.issue-4 |
| [33] | ROUX D L, ROOT B E, REEDY C R, et al. DNA analysis using an integrated microchip for multiplex PCR amplification and electrophoresis for reference samples[J]. Analytical Chemistry, 2014, 86(16): 8192–8199. DOI:10.1021/ac501666b |
| [34] | 季旭, 刘理天. 一种集成PCR反应室与CE的生物芯片的研究[J]. 仪器仪表学报, 2003(S2): 201–202. |
| [35] | LIU P, LI X, GREENSPOON S A, et al. Integrated DNA purification, PCR, sample cleanup, and capillary electrophoresis microchip for forensic human identification[J]. Lab on A Chip, 2011, 11(6): 1041–1048. DOI:10.1039/c0lc00533a |
| [36] | LE R D, ROOT B E, HICKEY J A, et al. An integrated sample-in-answer-out microfluidic chip for rapid human identification by STR analysis[J]. Lab on A Chip, 2014, 14(22): 4415–4425. DOI:10.1039/C4LC00685B |
| [37] | JOVANOVICH S, BOGDAN G, BELCINSKI R, et al. Developmental validation of a fully integrated sample-to-profile rapid human identification system for processing single-source reference buccal samples[J]. Forensic Science International Genetics, 2015, 16(1): 181–194. |
| [38] | HOLLAND M, WENDT F. Evaluation of the RapidHITTM 200, an automated human identification system for STR analysis of single source samples[J]. Forensic Science International Genetics, 2015(14): 76–85. |
| [39] | DATECHNONG M, HUDLOW W R, BUONCRISTIANI M R. Evaluation of the RapidHITTM 200 and RapidHIT GlobalFilerR Express kit for fully automated STR genotyping[J]. Forensic Science International Genetics, 2016, 23: 1–8. DOI:10.1016/j.fsigen.2016.03.001 |
| [40] | TAN E, TURINGAN R S, HOGAN C, et al. Fully integrated, fully automated generation of short tandem repeat profiles[J]. Investigative Genetics, 2013, 4(1): 16. DOI:10.1186/2041-2223-4-16 |
 2017, Vol. 43
2017, Vol. 43